Età biologica e fragilità: il ruolo della metilazione del DNA

In questa revisione sistematica/meta-analisi è stata esaminata l’associazione tra l’età biologica, misurata attraverso i cosiddetti “orologi di metilazione del DNA”, e la condizione di “frailty” o “fragilità” (1, 2). La ricerca bibliografica è stata condotta utilizzando sei database (Embase, Cochrane Central Register of Controlled Trials, PubMed, Ovid, Scopus e Web of Science), in un periodo compreso tra il 1° gennaio 2011 e il 6 giugno 2025, seguendo le linee guida PRISMA.
Quali studi sono stati presi in considerazione? Come sono state condotte le analisi dei dati?
Sono stati inclusi gli studi di coorte di popolazione sottoposti a revisione paritaria che coinvolgevano adulti di età media o mediana ≥18 anni provenienti da popolazioni generali o popolazioni specifiche per malattia contro un gruppo di controllo.
Sono stati considerati eleggibili gli studi trasversali o longitudinali, che valutavano le associazioni tra l’età di metilazione del DNA e la “fragilità”, quantificata utilizzando misure convalidate, quali l’indice di “fragilità” o il fenotipo di “fragilità” (2-4). È stata presa in considerazione l’età di metilazione del DNA ottenuta da metodi di raccolta di campioni minimamente invasivi (ad esempio, da campioni di sangue, saliva, cellule buccali o pelle) e sono stati considerati solo gli articoli che includevano risultati o predittori specificamente correlati agli orologi di metilazione del DNA, come l’età di metilazione del DNA, l’accelerazione dell’età di metilazione del DNA (nota anche come EAA) o le misurazioni della deviazione dell’età di metilazione (5).
Va ricordato come l’età di metilazione del DNA sia una misura diretta dell’età cronologica dell’individuo, mentre la deviazione dell’età e l’accelerazione dell’età di metilazione del DNA si riferiscono alla discrepanza tra l’età anagrafica di una persona e la sua età biologica o percepita e al delta di tale discrepanza, indicando così un invecchiamento accelerato.
Per ciascun orologio di metilazione del DNA sono state eseguite meta-analisi stratificate per la popolazione generale e specifica di malattia e sono state condotte standardizzazioni per consentire la comparabilità dei dati raccolti nei vari studi.
Ma cosa si intende col termine “orologio di metilazione del DNA”? Inoltre, in cosa consiste la “frailty” o “fragilità”?
Gli orologi di metilazione del DNA (o DNA methylation-based clocks) sono considerati biomarcatori dell’invecchiamento in quanto basati sul grado di metilazione del genoma (o DNA) (1, 2), un processo epigenetico che, aggiungendo gruppi metilici al DNA, regola l’attivazione e lo spegnimento dei geni, senza alterare la sequenza del DNA stesso. Avendo la capacità di intercettare l’invecchiamento accelerato a livello subcellulare, essi potrebbero, quindi, stimare l’età biologica effettiva di una persona e riflettere meglio la sua vulnerabilità fisica ed eventuale maggiore propensione a contrarre malattie, indipendentemente dagli anni che compaiono sul certificato di nascita. Per i suddetti motivi potrebbero essere utilizzati per identificare gli individui a rischio e consentire interventi geroprotettivi precoci.
Gli orologi di metilazione del DNA vengono suddivisi in:
- Orologi di prima generazione: si basano sull’età cronologica. Esempi di questi orologi includono Horvath1, Hannum, Horvath2, Lin, EpiTOC, 7-CpG e Mono-DNAmAge (6-9).
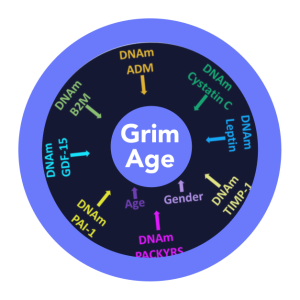
- Orologi di seconda generazione: si basano sugli esiti di salute (health outcomes). Esempi sono rappresentati da PhenoAge, GrimAge, GrimAge2, Zhang, DNAmFitAge e DNAmTL.(10-14)
- Orologi di terza generazione: si basano sul ritmo di invecchiamento (pace of ageing) Questi includono DunedinPoAm e DunedinPACE (15, 16).
- Orologi di quarta generazione: si basano sull’interpretabilità biologica. Esempi sono PRC2-AgeIndex, gli orologi arricchiti di causalità (causality-enriched clocks) e System Age (17-19).
La “frailty” o “fragilità” è una condizione età-correlata caratterizzata da una dis-regolazione e un declino fisiologico multi-sistemici. Tale condizione aumenta la vulnerabilità di un individuo a esiti sanitari avversi e lo espone a un maggior rischio di cadute, ospedalizzazione, disabilità e mortalità. Secondo i dati di letteratura, l’invecchiamento accelerato a livello cellulare e subcellulare rappresenterebbe il meccanismo causale alla base della suddetta dis-regolazione multisistemica che porta alla “frailty” (1,2,4).
Quale è stato quindi il risultato della ricerca bibliografica?
Sono stati identificati 34.437 articoli, dei quali 19.701 presenti in duplicato e quindi rimossi, mentre altri 13.206 sono stati esclusi in base ai titoli e all’abstract. Dei 1.211 articoli in forma di testo completo, 1.187 sono stati esclusi poiché utilizzavano uno strumento di quantificazione della “fragilità” non validato. I restanti 24 articoli che riportavano l’associazione tra metilazione del DNA e fragilità sono stati quindi inclusi nella revisione sistematica e 12 di questi sono stati considerati per la meta-analisi. Degli studi inclusi, 12 erano stati condotti in Europa, dieci in Nord America, due in Oceania e uno in Asia. Il campione totale includeva 28.325 individui (52,1% donne; età mediana 65,2 anni), con dimensioni del campione che andavano da 31 a 4.018 individui. Dei 24 articoli, 20 sono stati condotti nella popolazione generale (uno di questi focalizzato negli ultracentenari), mentre i restanti quattro sono stati condotti in popolazioni specifiche per malattia.
La “fragilità” è stata misurata prevalentemente utilizzando: l’indice di fragilità (15 studi) (20-22) o versioni adattate dell’indice di fragilità (tre studi) (23, 24), il fenotipo di fragilità (otto studi) (20, 25, 26), e altri indicatori come l’indice prognostico multidimensionale (27), l’indicatore di fragilità di Tilburg (27) e la scala di fragilità di Edmonton (28).
I risultati ottenuti mostrano come non in tutti gli studi condotti sia stato possibile evidenziare una correlazione tra gli orologi di metilazione del DNA di prima generazione e la “fragilità”. Questi orologi, infatti, essendo focalizzati sull’età cronologica, potrebbero non catturare la dis-regolazione multisistemica sottostante e il declino della riserva fisiologica (1, 29). Al contrario, gli orologi di metilazione del DNA di seconda generazione (PhenoAge, GrimAge e GrimAge2) e di terza generazione, che sono relativi al ritmo dell’invecchiamento (DunedinPoAm e DunedinPACE) e che integrano surrogati molecolari di parametri correlati alla salute e al rischio di mortalità (30-33), hanno rivelato associazioni positive più coerenti con la “fragilità”. Tra questi, in particolare, l’orologio di metilazione GrimAge risulta più coerentemente associato alla suddetta condizione.
E’ interessante sottolineare come il GRimAge rifletta il carico infiammatorio che contribuisce allo stato di “fragilità”, in quanto incorpora surrogati di metilazione del DNA correlati all’infiammazione (valutata mediante il dosaggio di PAI-1 e proteina C-reattiva [CRP]) (8).
Nessuno studio ha esaminato l’associazione tra gli orologi di metilazione del DNA di quarta generazione e la “fragilità”. Va ricordato come questi orologi si pongano l’obiettivo di identificare biomarcatori dell’invecchiamento non esclusivamente correlati con l’età cronologica o gli esiti di salute, ma che siano specificamente legati a processi biologici fondamentali e ai meccanismi sottostanti l’invecchiamento. Per questo motivo, rispetto agli orologi di prima-terza generazione, gli orologi di quarta generazione sono considerati più promettenti in quanto potenzialmente in grado di rilevare meglio il danno legato all’invecchiamento (ageing-related damage) e le risposte adattative (adaptive responses). Per esempio, è stato dimostrato che YingDamAge è in grado di intercettare gli effetti della rimozione del danno (damage-removal effects) indotti della riprogrammazione delle cellule staminali pluripotenti causata da una maggiore accelerazione dell’Età Epigenetica (EAA) in sindromi correlate all’invecchiamento e nell’epidermide esposta a luce ultravioletta.
In conclusione, i risultati ottenuti dalla revisione sistematica evidenziano quindi l’importanza dell’integrazione dei parametri di salute con quelli di metilazione del DNA al fine di migliorare le capacità degli orologi di identificare la condizione di “fragilità”.
Quali sono i limiti di questa revisione sistematica?
I limiti sono relativi alla tipologia di popolazione considerata, alla inclusione di prodotti bibliografici che utilizzavano definizioni di fragilità non sempre standardizzate e alla tipologia di approccio metodologico per la misurazione dei pattern di metilazione del DNA, che non sempre era quello più adeguato.
La generalizzabilità dei risultati è stata, ad esempio, inficiata dall’avere avere incluso pochi studi condotti nella popolazione anziana (≥80 anni), che è quella più vulnerabile e a rischio di maggiore “fragilità”. Inoltre, la “frailty” è stata quantificata utilizzando strumenti diversi, tra cui l’indice di fragilità e il fenotipo di fragilità, ciascuno dei quali in grado di “catturare” aspetti differenti di tale condizione (2,3).
Il fatto di avere incluso campioni eterogenei tra di loro e di avere considerato un numero limitato di studi non ha quindi reso possibile trarre conclusioni definitive e generalizzabili nella popolazione.
Quali sono le possibili azioni correttive volte a migliorare la generalizzabilità dei risultati?
Andrebbe sicuramente aumentata la numerosità del campione, e bisognerebbe includere popolazioni diverse, di grandi anziani, di etnia e background socioeconomici diversi. Sarebbe necessario, inoltre, standardizzare i metodi di quantificazione dei pattern di metilazione del DNA e di quantificazione della “fragilità”. Un aspetto cruciale che andrebbe affrontato è quello relativo all’approfondimento dei percorsi meccanicistici che collegano le alterazioni epigenetiche alle manifestazioni cliniche della “frailty”. In quest’ottica, ricerche future potrebbero, ad esempio, essere volte a chiarire se l’accelerazione dei processi di metilazione del DNA può contribuire allo sviluppo della fragilità o se è la fragilità stessa che è in grado di accelerare la metilazione del DNA. Infine, studi interventistici mirati a determinanti modificabili delle alterazioni della metilazione del DNA potrebbero essere fondamentali per tradurre questi risultati nella pratica clinica.
Bibliografia
- Fried LP, Cohen AA, Xue Q-L, Walston J, Bandeen-Roche K, Varadhan R. The physical frailty syndrome as a transition from homeostatic symphony to cacophony. Nat Aging 2021; 1: 36–46.
- Fried LP, Tangen CM, Walston J, et al. Frailty in older adults:evidence for a phenotype. J Gerontol A Biol Sci Med Sci 2001; 56: M146–56.
- Kim DH, Rockwood K. Frailty in older adults. N Engl J Med 2024; 391: 538–48. 4. Wang XM, Zhang YH, Meng CC, et al. Scale-based screening and assessment of age-related frailty. Front Public Health 2024; 12: 1424613.
- Teschendorff AE, Horvath S. Epigenetic ageing clocks: statistical methods and emerging computational challenges. Nat Rev Genet 2025; 26: 350–68.
- Moqri M, Herzog C, Poganik JR, et al. Biomarkers of aging for the identification and evaluation of longevity interventions. Cell 2023; 186: 3758–75.
- Bell CG, Lowe R, Adams PD, et al. DNA methylation aging clocks: challenges and recommendations. Genome Biol 2019; 20: 249.
- Morandini F, Rechsteiner C, Perez K, et al. ATAC-clock: an aging clock based on chromatin accessibility. Geroscience 2024; 46: 1789–806.
- de Lima Camillo LP, Asif MH, Horvath S, Larschan E, Singh R. Histone mark age of human tissues and cell types. Sci Adv 2025; 11: eadk9373.
- Levine ME, Lu AT, Quach A, et al. An epigenetic biomarker of aging for lifespan and healthspan. Aging 2018; 10: 573–91.
- Lu AT, Quach A, Wilson JG, et al. DNA methylation GrimAge strongly predicts lifespan and healthspan. Aging 2019; 11: 303–27.
- Lu AT, Binder AM, Zhang J, et al. DNA methylation GrimAge version 2. Aging 2022; 14: 9484–549.
- Zhang Y, Wilson R, Heiss J, et al. DNA methylation signatures in peripheral blood strongly predict all-cause mortality. Nat Commun 2017; 8: 14617.
- McGreevy KM, Radak Z, Torma F, et al. DNAmFitAge: biological age indicator incorporating physical fitness. Aging 2023; 15: 3904–38.
- Lu AT, Seeboth A, Tsai PC, et al. DNA methylation-based estimator of telomere length. Aging 2019; 11: 5895–923.
- Belsky DW, Caspi A, Arseneault L, et al. Quantification of the pace of biological aging in humans through a blood test, the DunedinPoAm DNA methylation algorithm. eLife 2020; 9: e54870.
- Belsky DW, Caspi A, Corcoran DL, et al. DunedinPACE, a DNA methylation biomarker of the pace of aging. eLife 2022; 11: e73420.
- Moqri M, Cipriano A, Simpson DJ, et al. PRC2-AgeIndex as a universal biomarker of aging and rejuvenation. Nat Commun 2024; 15: 5956.
- Ying K, Liu H, Tarkhov AE, et al. Causality-enriched epigenetic age uncouples damage and adaptation. Nat Aging 2024; 4: 231–46.
- Sehgal R, Markov Y, Qin C, et al. Systems Age: a single blood methylation test to quantify aging heterogeneity across 11 physiological systems. bioRxiv 2024; published online May 28. https://doi.org/10.1101/2023.07.13.548904 (preprint).
- Kim S, Myers L, Wyckoff J, Cherry KE, Jazwinski SM. The frailty index outperforms DNA methylation age and its derivatives as an indicator of biological age. Geroscience 2017; 39: 83–92.
- Kuiper LM, Polinder-Bos HA, Bizzarri D, et al. Epigenetic and metabolomic biomarkers for biological age: a comparative analysis of mortality and frailty risk. J Gerontol A Biol Sci Med Sci 2023; 78: 1753–62.
- Li X, Ploner A, Wang Y, et al. Longitudinal trajectories, correlations and mortality associations of nine biological ages across 20-years follow-up. eLife 2020; 11: e51507.
- LaHue SC, Fuentealba M, Roa Diaz S, et al. Association of biological aging with frailty and post-transplant outcomes among adults with cirrhosis. Geroscience 2023; 46: 3287–95.
- Liang X, Aouizerat BE, So-Armah K, et al. DNA methylation-based telomere length is associated with HIV infection, physical frailty, cancer, and all-cause mortality. Aging Cell 2024; 23: e14174.
- Gale CR, Marioni RE, Harris SE, Starr JM, Deary IJ. DNA methylation and the epigenetic clock in relation to physical frailty in older people: the Lothian Birth Cohort 1936. Clin Epigenetics 2018; 10: 101.
- García-delaTorre P, Rivero-Segura NA, Sánchez-García S, et al. GrimAge is elevated in older adults with mild COVID-19 an exploratory analysis. Geroscience 2024; 46: 3511–24.
- Kuiper LM, Polinder-Bos HA, Bizzarri D, et al. Epigenetic and metabolomic biomarkers for biological age: a comparative analysis of mortality and frailty risk. J Gerontol A Biol Sci Med Sci 2023; 78: 1753–62.
- Safaee MM, Dwaraka VB, Lee JM, et al. Epigenetic age biomarkers and risk assessment in adult spinal deformity: a novel association of biological age with frailty and disability. J Neurosurg Spine 2024; 40: 312–23.
- Oblak L, van der Zaag J, Higgins-Chen AT, Levine ME, Boks MP. A systematic review of biological, social and environmental factors associated with epigenetic clock acceleration. Ageing Res Rev 2021; 69: 101348.
- Levine ME, Lu AT, Quach A, et al. An epigenetic biomarker of aging for lifespan and healthspan. Aging 2018; 10: 573–91.
- Lu AT, Quach A, Wilson JG, et al. DNA methylation GrimAge strongly predicts lifespan and healthspan. Aging 2019; 11: 303–27.
- Lu AT, Binder AM, Zhang J, et al. DNA methylation GrimAge version 2. Aging 2022; 14: 9484–549.
- Zhang Y, Wilson R, Heiss J, et al. DNA methylation signatures in peripheral blood strongly predict all-cause mortality. Nat Commun 2017; 8: 14617



